È passato quasi un anno dall’inizio dell’epidemia di COVID-19. Partito dalla città di Wuhan, in Cina, il coronavirus SARS-CoV-2 ha infettato oltre 50 milioni di persone nel mondo, uccidendone il 2.5% (dati Johns Hopkins). Gli scienziati sono in larga parte convinti che il virus abbia un’origine naturale, ma sono molte le cose che ancora non sappiamo. Le analisi genetiche ci dicono chiaramente che l’antenato di SARS-CoV-2 era un virus dei pipistrelli, con ogni probabilità originario della Cina meridionale: entrambi i suoi parenti più prossimi (RaTG13 e RmYN02) sono stati identificati infatti nella provincia dello Yunnan, così come moltissimi altri virus di tipo SARS finora scoperti nei pipistrelli. Non è ancora chiaro, invece, in che modo esattamente questo virus abbia percorso i 1000 km che separano lo Yunnan dalla città di Wuhan, sede dell’Istituto di Virologia che da anni si occupa proprio di studiare i coronavirus presenti nei pipistrelli. Su questo punto le opinioni divergono, anche all’interno della stessa comunità scientifica.
Per molti il virus è passato all’uomo attraverso un ospite intermedio: un animale presumibilmente in vendita in un wet market, uno di quei contesti dove uomini e fauna selvatica vengono a trovarsi a più stretto contatto. Dopo tutto era avvenuto proprio questo con la prima SARS, che aveva fatto il “salto” dagli zibetti all’uomo, e con la MERS, che era passata attraverso i cammelli. Ma l’animale questa volta non si trova: inizialmente si è pensato al mercato del pesce di Huanan, ma le analisi degli scienziati cinesi hanno trovato tracce del virus solo sulle superfici e non negli animali in vendita. Le ricerche sono poi proseguite in tutta la provincia dello Hubei, andando a campionare il bestiame allevato nelle fattorie, anche questa volta senza successo. Nel caso della prima SARS le cose erano andate diversamente: il primo caso apparve nel Guangdong nel novembre 2002, il patogeno fu identificato ad aprile 2003 e già a maggio avevamo scoperto negli zibetti in vendita nei mercati una versione quasi identica del virus (Guan et al., 2003). Per certi versi, era una situazione opposta rispetto a quella attuale: non sapevamo ancora che i pipistrelli erano l’animale serbatoio, ma sono stati sufficienti sei mesi per trovare l’ospite intermedio. Viste le difficoltà che stiamo incontrando questa volta, gli scienziati stanno prendendo in considerazione anche un’altra possibilità, più remota e molto complessa da dimostrare: il virus potrebbe avere fatto il salto (lo spillover) direttamente dai pipistrelli, e poi essersi evoluto lentamente tra gli esseri umani, adattandosi sempre più al nuovo ospite nel corso del tempo. In questo caso, ci si aspetterebbe di trovare tracce del passaggio del virus anche in altre regioni della Cina, prima dello scorso dicembre: ad esempio, casi sporadici di polmoniti atipiche causate da un virus simile a SARS-CoV-2. Ma se queste polmoniti ci sono state, per quel che ne sappiamo le autorità sanitarie cinesi non le hanno rilevate.
Resta una terza, inquietante, possibilità, che solo pochi scienziati si rifiutano di escludere: quella che SARS-CoV-2 sia uscito per errore da un laboratorio, e che, addirittura, possa essere il risultato di manipolazioni genetiche. Questa ipotesi sarebbe per altro compatibile con il fatto che il virus è sembrato già ben adattato all’uomo nel momento in cui è apparso a Wuhan, come suggerito da un preprint di maggio e ora confermato anche dall’OMS, che pure crede all’origine naturale. I nomi degli scienziati che ritengono realistico questo scenario sono pochissimi, o almeno sono pochissimi quelli che sostenuto questa posizione pubblicamente: penso al biologo Richard Ebright, all’immunologo Nikolai Petrovskj, al virologo Étienne Decroly, al microbiologo David Relman e alla ricercatrice Alina Chan del Broad Institute, ormai una star su Twitter, la quale da sempre sostiene che, sulla base delle evidenze disponibili, non si possa escludere nessuno scenario. Il fatto è che qualsiasi discorso sulle origini del virus si scontra inevitabilmente con questioni politiche. Fin da subito Donald Trump ha accusato la Cina di aver creato SARS-CoV-2 in uno dei suoi laboratori, e ho la sensazione che il dibattito all’interno della comunità scientifica sia inquinato da pressioni politiche. La tentazione di attribuire un’etichetta politica a chiunque prenda in considerazione, anche solo lontanamente, l’ipotesi del laboratorio è molto forte, e di questi tempi sono pochissimi gli scienziati che vogliono essere accostati a Trump (come dar loro torto!). Resta quindi il dubbio che l’ipotesi dell’origine naturale sia stata spinta, oltre che dalle evidenze, anche (e forse di più) da un sentimento di avversione nei confronti del Presidente americano uscente, e dalla paura di alimentare la sua narrazione.
In questi mesi ho seguito molto da vicino la questione delle origini del virus, ho letto moltissimo e scoperto cose sorprendenti. Dopo tanto tempo, ho deciso di condividere con voi tutto quello che ho imparato, fornendovi tutti i riferimenti e i link che possano servirvi per valutare la correttezza di quanto affermo. Non mi dilungherò parlando di improbabili teorie complottiste come quella di Li-Meng Yan (altri ne hanno già scritto), mi concentrerò invece sui dati e sulla letteratura scientifica ufficiale, e vi assicuro che sarà un racconto interessante. Al termine di questo lunghissimo articolo, saprete (quasi) tutto quello che c’è da sapere sulle origini di questo virus, ma soprattutto quello che ancora non sappiamo.
Mattoncini Lego e furetti con l’influenza
Se vogliamo valutare in modo serio l’ipotesi del laboratorio, la prima domanda a cui dovremmo dare una risposta è: con le attuali conoscenze scientifiche e tecnologiche, è possibile costruire un virus in laboratorio? Si tratta di un requisito ovvio, quasi banale, ma è bene discuterne, per chiarire quello che si può e quello che, invece, non si può fare. L’ingegneria genetica ha fatto passi da gigante negli ultimi anni, basti pensare al premio Nobel ricevuto poche settimane fa da Emmanuelle Charpentier e Jennifer Doudna, coinventrici della tecnica di genome editing nota come CRISPR/Cas9. Siamo in grado di modificare il genoma di batteri, piante e animali (uomini, persino) con molta precisione, inserendo pezzi di DNA di altri organismi, come è stato fatto per anni con gli OGM, o anche sostituendo singole “lettere” del genoma. Da questo punto di vista, per quanto riguarda i virus la situazione non è molto diversa, anzi: avendo dimensioni ridotte, i genomi virali possono essere rimaneggiati e assemblati quasi come se fossero dei mattoncini Lego. Come vedremo, anche la tecnica dei Lego si è evoluta e affinata negli ultimi tempi, ma parliamo comunque di metodi di biologia molecolare che sono utilizzati almeno dagli anni 90: Ralph Baric, probabilmente il maggior esperto al mondo di coronavirus, aveva costruito già nel 2002 un “clone” sintetico del virus dell’epatite murina in grado di infettare cellule di criceto (Young et al., 2002). E pochi mesi fa, alcuni scienziati dell’Università di Berna sono riusciti a ricreare il virus SARS-CoV-2 nel giro di una settimana, assemblando frammenti del genoma virale che avevano ordinato da un’azienda specializzata in sintesi di DNA e proteine (Thao et al, 2020). Con le tecniche e le conoscenze attuali, sarebbe quindi teoricamente possibile sintetizzare un virus (o meglio, un clone virale) che, trasferito in cellule appropriate, sia in grado di replicarsi proprio come un virus naturale. Come vedremo, è anche possibile ricombinare tra loro pezzi di virus differenti, creando in questo modo le cosiddette “chimere”. Si chiama reverse genetics (o “genetica inversa”), e come abbiamo visto non è neppure necessario avere a disposizione i virus originali da ricombinare; basta conoscere la sequenza di DNA e ordinarne la sintesi da una società specializzata.
Esiste poi un altro modo, meno sofisticato, per generare virus nuovi in laboratorio. Se si prende un virus completamente naturale e lo si fa crescere in colture cellulari, è possibile in un certo senso indirizzarne l’evoluzione, andando a selezionare quelle mutazioni che permettono al virus di replicarsi più efficacemente nello specifico tipo di cellule con cui viene messo a contatto. In questo caso non stiamo più parlando di ingegneria genetica in senso stretto: qui assistiamo a meccanismi evolutivi che potrebbero avvenire tranquillamente in natura, con la differenza che in laboratorio, fornendo le condizioni ottimali, possiamo “accelerare” questi processi. Già nel 1997, un paper pubblicato sul Journal of Virology spiegava come fosse possibile (con molta pazienza) mutare del 2% la proteina spike di un coronavirus di topo, semplicemente trasferendo le cellule infette da una piastra all’altra per 600 volte (Schickli et al., 1997). La stessa tecnica si può usare, oltre che sulle cellule, anche sugli animali da laboratorio: è celebre l’esperimento del gruppo di Ron Fouchier, che nel 2012 rese il letale virus dell’influenza aviaria H5N1 trasmissibile tra furetti per via aerea, attraverso diversi passaggi di infezione sugli animali (Herfst et al., 2012).
Abbiamo quindi a disposizione diverse tecniche per modificare e ricombinare i virus in laboratorio, alcune più sofisticate e altre meno. Quello che invece, ancora oggi, è difficilissimo fare è generare un virus completamente nuovo: l’atto “creativo” di inventare da zero una sequenza che possa dare vita a un virus funzionante è ancora al di fuori della nostra portata. Certo, negli ultimi anni abbiamo maturato moltissime conoscenze circa i meccanismi molecolari di infezione dei virus: una review dell’anno scorso, firmata proprio da ricercatori dell’Istituto di Virologia di Wuhan, elenca con precisione gli amminoacidi che permettono ai coronavirus di tipo SARS di legarsi al recettore umano ACE2 (Cui et al., 2019). E non dimentichiamoci che oggi abbiamo anche il supporto dei computer: grazie alle simulazioni, possiamo testare l’effetto di una nuova mutazione prima ancora di inserirla nel genoma di un virus. In tutti i casi, però, si tratta sempre di replicare soluzioni già viste in natura. Che si tratti di cambiare una singola lettera nel genoma di un virus noto, o assemblarne uno nuovo rimescolando pezzi di virus diversi, il punto di partenza è sempre il genoma (o parti di genoma) di virus già esistenti in natura. Un’altra cosa che è molto difficile fare è prevedere cosa accadrà al virus una volta rilasciato nell’ambiente: un conto è rimodellare la proteina spike di un coronavirus in modo che sia in grado di legarsi meglio al recettore umano, un altro è controllare in modo preciso i sintomi che questo virus mutato scatenerà negli uomini al di fuori del laboratorio, così come la modalità e la velocità di trasmissione da un soggetto a un altro. È quindi molto improbabile che le caratteristiche che rendono così efficace SARS-CoV-2 siano state disegnate a tavolino in qualche laboratorio militare segreto.
Fatta questa dovuta premessa tecnologica, è ora il momento di entrare più a fondo nella questione delle origini, andando a scoprire quali esperimenti si conducevano nei laboratori di Wuhan, la capitale dello Hubei da cui tutto è iniziato. Se costruire un virus sintetico è – almeno in linea di principio – un’operazione alla portata di molti laboratori, diverso è dimostrare che nei laboratori di quella città queste attività erano effettivamente svolte, e se sì per quale motivo.
Pipistrelli e chimere
Dall’emergenza SARS del 2002, l’Istituto di Virologia di Wuhan è uno dei centri di ricerca in prima linea nella prevenzione delle epidemie. La missione scientifica dell’istituto, e in particolare del team coordinato dalla virologa Shi Zhengli, è, infatti, quella di scovare nella fauna selvatica i coronavirus potenzialmente in grado di fare il famoso spillover ed eventualmente scatenare una pandemia. Alle ricerche del centro di Wuhan hanno contribuito anche gli Stati Uniti, soprattutto attraverso cospicui finanziamenti per l’organizzazione non governativa EcoHealth Alliance, guidata da Peter Daszak, che con i ricercatori di Wuhan – e in particolare con la professoressa Shi Zhengli – ha stretto una lunga e proficua collaborazione scientifica (si vedano i grant NIH: 1R01AI110964-01, 2R01AI110964-06).
I primi studi della prof.ssa Shi si concentrano sulla proteina spike, l’arpione molecolare che consente ai coronavirus di tipo SARS di agganciarsi ai recettori ACE2 delle nostre cellule. Testando varie combinazioni di amminoacidi con la tecnica dei mattoncini Lego descritta più sopra, gli scienziati cinesi scoprono la regione chiave dello spike, quella che – se correttamente mutata – permetteva ai coronavirus dei pipistrelli di infettare le cellule umane (Ren et al., 2008). Negli anni seguenti, Shi Zhengli si mette alla ricerca dell’antenato della SARS, esplorando le campagne della Cina meridionale, note per essere popolate da diverse specie di pipistrelli. Nel 2013 pubblica su Nature la scoperta di due virus dei pipistrelli molto simili alla SARS; in particolare, uno dei due (ribattezzato WIV1) era in grado di crescere in colture cellulari di mammifero e di riconoscere il nostro recettore ACE2 (Ge et al., 2013). Le capacità del secondo virus (SHC014) erano invece meno chiare, pertanto i ricercatori decidono di proseguire gli studi coinvolgendo lo scienziato americano Ralph Baric, uno dei maggiori esperti mondiali nella costruzione di virus sintetici. Scopo di questa collaborazione è capire se lo spike di SHC014 abbia già le carte in regola per fare il salto di specie. Per scoprirlo, Shi e Baric costruiscono una chimera: lo spike di SHC014 viene montato su un virus della SARS adattato ai topi chiamato MA1, ottenuto in laboratorio da Baric diversi anni prima (Roberts et al., 2007). Testata su cellule umane, la chimera mostrava effetti simili a quelli della SARS, dimostrando quindi che anche lo spike di questo virus aveva già le caratteristiche giuste per attaccare l’uomo. Non doveva adattarsi al recettore umano insomma, aveva solamente bisogno del contesto genomico corretto, che in questo caso era fornito dal virus MA1 (Menachery et al., 2015).
Le ricerche di Shi Zhengli, ormai nota come “Bat Woman”, continuano. Nel 2016 annuncia il ritrovamento di un coronavirus di tipo SARS in una miniera abbandonata nello Yunnan (Ge et al., 2016), mentre l’anno successivo pubblica le sequenze di ben 11 nuovi coronavirus di tipo SARS identificati sempre nello Yunnan tra il 2011 e il 2015. Lo studio, apparso su PLoS Pathogens, ha grande risonanza a livello internazionale, perché proprio dalla ricombinazione di questi virus sembra essersi originata la prima SARS. Ai fini del nostro racconto, è interessante notare che in questo lavoro costruiscono 8 diverse chimere, inserendo lo spike di questi nuovi virus dei pipistrelli nel genoma di WIV1, il primo virus isolato dall’istituto nel 2013. Due di queste chimere sono in grado di infettare cellule umane, dimostrando ancora una volta la presenza nei pipistrelli di coronavirus già pronti per il salto di specie (Hu et al., 2017). L’ipotesi è ulteriormente confermata da uno studio del 2018 della stessa Shi Zhengli: in un’indagine sierologica condotta nel 2015 tra abitanti dello Yunnan che vivevano in prossimità di grotte popolate da pipistrelli, i ricercatori scoprono la presenza di anticorpi anti-SARS in 6 persone sulle 218 testate, evidenza del fatto che lo spillover dai pipistrelli all’uomo, benché sporadicamente, poteva effettivamente avvenire (Wang et al., 2018). Grazie alle ricerche della Bat Woman abbiamo imparato molte cose sui coronavirus e sugli spillover: conosciamo le zone geografiche da monitorare (come lo Yunnan), le specie di pipistrelli portatrici dei virus più pericolosi e persino i singoli amminoacidi della proteina spike che è necessario mutare per permettere il salto di specie (Yu et al., 2019; Fan et al., 2019; Cui et al., 2019).
Il lavoro di Shi Zhengli – lo avrete notato – si componeva essenzialmente di due parti: da un lato, la ricerca sul campo, con le esplorazioni delle grotte del sud della Cina e la raccolta di campioni dei pipistrelli; dall’altro, la ricerca in laboratorio, con gli esperimenti di infezione per verificare la pericolosità dei virus trovati in natura. In questa seconda parte del lavoro si utilizzavano anche tecniche di ingegneria genetica, in particolare si costruivano chimere, essenzialmente per testare gli spike dei nuovi coronavirus scoperti nei pipistrelli. A questo punto, resta una sola fondamentale domanda a cui dobbiamo dare una risposta: se nell’ambito delle loro ricerche gli scienziati di Wuhan avessero costruito SARS-CoV-2, combinando tra loro virus dei pipistrelli, noi ce ne accorgeremmo? In altre parole: se fosse un virus artificiale, si vedrebbe?
Scheletri e cicatrici
In questi mesi vi sarà capitato di sentire o di leggere dichiarazioni come questa: “SARS-CoV-2 non è il prodotto di ingegneria genetica, perché se lo fosse si vedrebbe: ci sarebbero delle cicatrici“. Ma cosa sono esattamente queste cicatrici? Per rispondere dobbiamo parlare di nuovo della tecnica dei mattoncini Lego. Come scrivevo più sopra, da diversi anni i virologi sono in grado di realizzare virus sintetici combinando tra loro pezzi di DNA differenti. Siccome il genoma di un virus, per quanto piccolo, è comunque molto grande per essere sintetizzato per intero, la strategia utilizzata è infatti quella di ottenere separatamente i diversi frammenti che poi vengono riassemblati a generare il genoma completo. Ogni frammento è inserito all’interno di un plasmide, ossia una molecola di DNA circolare che funge da supporto; con l’aiuto di appositi enzimi, i ricercatori staccano il frammento che interessa e lo agganciano agli altri. Le “cicatrici” di cui si parla si vengono a formare proprio durante questo passaggio: poiché gli enzimi in questione riconoscono specifiche sequenze di taglio, quando i frammenti vengono poi assemblati rimane traccia, nelle giunzioni tra un frammento e l’altro, di queste sequenze segnale. Questo, almeno, era quello che accadeva fino a qualche tempo fa. Negli ultimi anni, infatti, sono state sviluppate tecniche di assemblaggio alternative, più sofisticate e versatili, che non lasciano più queste tracce. Si chiamano per l’appunto tecniche seamless (in inglese “senza cuciture”), ne esistono di vari tipi (Golden Gate Assembly, Gibson assembly) e molte di queste sono già confezionate in appositi kit commerciali acquistabili online. Le cicatrici di cui qualcuno parla, quindi, si vedrebbero sicuramente, ma solo se il virus fosse stato costruito molti anni fa. D’altra parte, lo stesso Ralph Baric, che di queste cose si occupa da sempre e di certo non sostiene l’origine artificiale del virus, ha dichiarato alla trasmissione Presa Diretta che, con i metodi di assemblaggio sviluppati negli ultimi anni, un virus sintetizzato in laboratorio sarebbe del tutto indistinguibile da uno naturale. Bene, dunque sulle cicatrici non possiamo più fare affidamento. Cosa ci resta?
Una motivazione ripetuta molto spesso e usata anche nel famoso articolo “The proximal origin of SARS-CoV-2” ha a che fare con la mancanza di un backbone noto, ossia dello “scheletro” di un virus già conosciuto (Andersen et al., 2020). Tale motivazione si basa su un presupposto corretto: come scrivevo all’inizio, per creare un virus nuovo è necessario partire da un virus esistente in natura. Se per assurdo costruissi una chimera combinando un pezzo del virus dell’influenza e un pezzo di SARS-CoV-2, e qualcuno lo trovasse e ne analizzasse il genoma, sarebbe sicuramente in grado di riconoscere i due virus di partenza e inizierebbe a domandarsi se una simile ricombinazione sarebbe possibile in natura o se invece potrebbe essere opera di ingegneria genetica. Vista così, la cosa ha perfettamente senso. Ma c’è un altro presupposto su cui si basa la motivazione del backbone noto, e questa volta è un presupposto sbagliato: l’idea che tutti i virus noti siano pubblici. Certo, esistono database pubblici in cui gli scienziati caricano le sequenze dei virus che stanno studiando, ma nessuno li obbliga a farlo. Qualcuno forse storcerà il naso davanti a questa affermazione, ma vi assicuro che non si tratta di un modo di ragionare complottista, e la dimostrazione è sotto gli occhi di tutti. Pochi mesi fa, l’Istituto di Virologia di Wuhan ha pubblicato la sequenza del genoma di RaTG13, il virus dei pipistrelli più simile a SARS-CoV-2 tra tutti quelli conosciuti. Per stessa ammissione di Shi Zhengli, però, conoscevano quel genoma già dal 2018, e il campione originale si trovava nei laboratori di Wuhan addirittura dal 2013. La prof.ssa Shi giura di non aver fatto altri esperimenti su RaTG13, sequenziamento a parte; ma se li avesse fatti e il virus fosse uscito dal laboratorio, qualcuno si sarebbe accorto della sua origine artificiale, oppure lo avrebbero etichettato come “naturale” perché non esisteva un backbone noto? Con queste riflessioni non intendo assolutamente sostenere che SARS-CoV-2 sia uscito da un laboratorio, non lo posso sapere e non ci sono prove che questo sia avvenuto. Quello che voglio dire è che l’analisi del genoma di questo virus non può darci le risposte che cerchiamo: non ci sono segni evidenti di ingegneria genetica, è vero, tuttavia l’assenza di evidenza non è, di per sé, evidenza di assenza. Ma se le manipolazioni genetiche non lasciano tracce, come possiamo scoprire la verità? Ci restano due alternative: la prima è trovare prove più convincenti dell’origine naturale (ad esempio il famoso animale ospite intermedio), la seconda è verificare che i ricercatori di Wuhan abbiano sempre agito in modo trasparente ed è quindi sciocco dubitare della loro buona fede. Questo non proverebbe nulla in modo definitivo, ma perlomeno allontanerebbe molti dei sospetti che aleggiano su quei laboratori.
Miniere e database
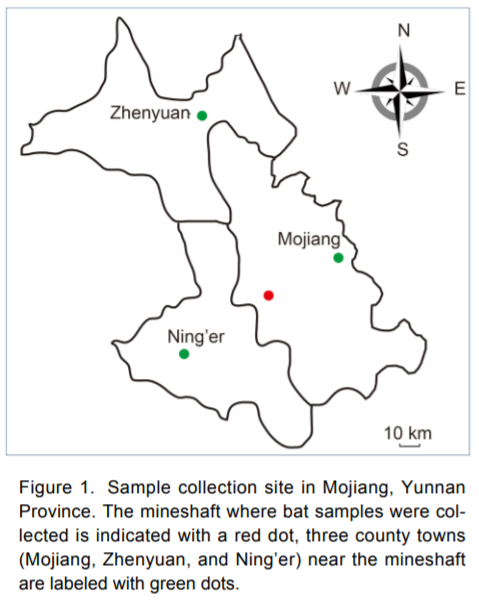
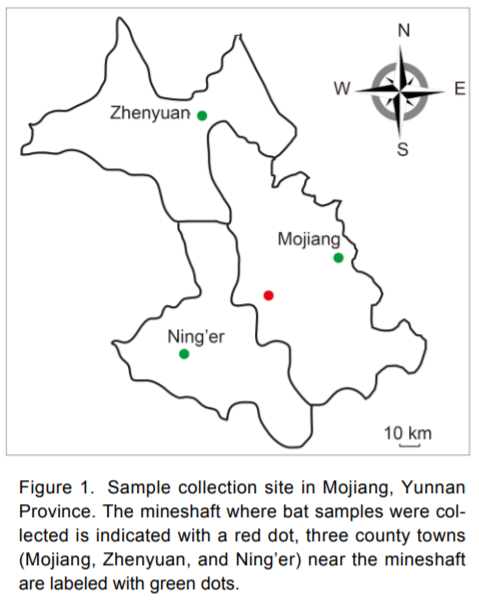
Tocca ammetterlo: quanto a trasparenza, i ricercatori cinesi non sono stati impeccabili. E forse è proprio questa mancanza di trasparenza il motivo per cui ancora oggi, dopo quasi un anno dallo scoppio dell’epidemia, continuano a circolare sui social ipotesi più o meno fantasiose su eventuali responsabilità degli scienziati di Wuhan. In particolare, i sospetti ruotano attorno a RaTG13, il virus dei pipistrelli più simile a SARS-CoV-2 tra quelli a oggi conosciuti. Il team di Shi Zhengli lo aveva trovato nel 2013 in una miniera di rame abbandonata della contea di Mojiang, nella provincia meridionale dello Yunnan. I ricercatori cinesi erano stati chiamati a indagare sulla vicenda di sei persone che erano entrate nella miniera, popolata da varie specie di pipistrelli, e avevano contratto delle polmoniti atipiche con sintomi molto simili a quelli di COVID-19 (Rahalkar et al, 2020). Gli scienziati avevano raccolto centinaia di campioni di feci di pipistrello, avevano estratto l’RNA e lo avevano analizzato, alla ricerca di virus simili alla SARS che potessero spiegare le polmoniti (tre dei pazienti erano morti). Come descritto in un articolo pubblicato tre anni dopo, avevano sequenziato una parte del gene RdRp e in questo modo avevano individuato 152 diversi coronavirus; di questi 152, due erano betacoronavirus, e soltanto uno era del tipo SARS (Ge et al., 2016). I ricercatori lo avevano trovato in un pipistrello a ferro di cavallo della specie Rhinolophus affinis, e lo avevano chiamato RaBtCov/4991. Da un punto di vista genetico, aveva caratteristiche diverse dagli altri coronavirus di tipo SARS noti fino a quel momento, tanto da essere considerato un possibile nuovo ceppo. Scrivono infatti gli autori:
In the phylogenetic tree, RaBtCoV/4991 showed more divergence from human SARS-CoV than other bat SL-CoVs and could be considered as a new strain of this virus lineage.
Ge et al., 2016 Virologica Sinica
All’inizio di quest’anno, i ricercatori dell’Istituto di Virologia di Wuhan hanno annunciato dalle pagine di Nature la scoperta di un virus dei pipistrelli con un’identità del 96% rispetto al nuovo coronavirus (Zhou et al., 2020). Si chiama RaTG13, ed era stato trovato – così scrivono – nella provincia dello Yunnan. Non si forniscono altri dettagli riguardo questo virus, che a tutti gli effetti è una novità assoluta nella letteratura scientifica. Non solo, ma nel paragrafo che descrive le analisi svolte su RaTG13 non sono esplicitate le tempistiche di questi esperimenti, lasciando intendere che il suo genoma sia stato sequenziato a inizio 2020, o al massimo a fine 2019 (in ogni caso dopo lo scoppio dell’epidemia). In questa versione dei fatti, gli scienziati confrontano il gene RdRp di RaTG13 con il gene corrispondente di SARS-CoV-2, notano una forte somiglianza e per questo motivo decidono di sequenziare per intero il genoma del vecchio virus campionato nello Yunnan.
We then found that a short region of RNA-dependent RNA polymerase (RdRp) from a bat coronavirus (BatCoV RaTG13)—which was previously detected in Rhinolophus affinis from Yunnan province—showed high sequence identity to 2019-nCoV. We carried out full-length sequencing on this RNA sample (GISAID accession number EPI_ISL_402131). Simplot analysis showed that 2019-nCoV was highly similar throughout the genome to RaTG13 (Fig. 1c), with an overall genome sequence identity of 96.2%.
Zhou et al., 2020 Nature
Gli autori non citano il lavoro del 2016 (firmato per altro da loro stessi), ma con un banale allineamento si scopre che il gene RdRp del “nuovo” RaTG13 è identico a quello del “vecchio” RaBtCov/4991 scoperto nella miniera dello Yunnan. Che i due nomi facciano riferimento allo stesso virus è stato poi confermato dalla stessa Shi Zhengli, in una delle risposte date quest’estate alla rivista Science. Non c’è nulla di scandaloso a cambiare nome a un vecchio virus, ma la scelta degli autori di non citare il vecchio studio del 2016 è certamente una stranezza: in genere, negli articoli scientifici si tende sempre a mettere una citazione in più, piuttosto che una in meno, soprattutto se quella citazione rimanda a un tuo lavoro. Il fatto che questa volta ciò non sia avvenuto ha destato qualche sospetto: la mancanza di quel link, infatti, ha reso più difficoltoso il collegamento con la storia della miniera e delle polmoniti. Il patogeno killer della famosa miniera non è mai stato identificato ufficialmente, benché la prof.ssa Shi abbia dichiarato a Scientific American che il responsabile fosse un fungo. In realtà, una tesi di laurea scovata da un utente Twitter (@TheSeeker) descrive molte delle indagini svolte su quella vicenda e conclude che un coronavirus di tipo SARS poteva effettivamente essere la causa di quelle polmoniti. A suggerirlo – si legge in quel documento – era niente meno che l’epidemiologo e pneumologo Zhong Nanshan, probabilmente il medico cinese più stimato al mondo. Alla luce di tutte queste informazioni, è ragionevole ipotizzare che i ricercatori di Wuhan, omettendo quella citazione nel loro articolo, abbiano cercato di nascondere il collegamento con una storia scomoda. Forse volevano semplicemente evitare che qualcuno li accusasse di essersi fatti sfuggire l’antenato di SARS-CoV-2, antenato che magari era presente anch’esso in quella miniera, insieme a RaTG13. Dopotutto, prevenire le epidemie era proprio la missione scientifica del gruppo di Shi Zhengli, e raccontare al mondo quella storia era un po’ come ammettere il proprio fallimento. Le omissioni nell’articolo di Nature, però, hanno portato qualcuno a sospettare scenari ancora peggiori: forse in quella miniera l’antenato di SARS-CoV-2 c’era davvero, gli scienziati di Wuhan lo hanno scoperto, portato nei loro laboratori e da lì è uscito per errore, magari dopo esperimenti e modifiche genetiche che – come abbiamo visto – non lasciano tracce. Si tratta solo di speculazioni, nulla di tutto ciò è dimostrato, ma certamente la mancanza di trasparenza non aiuta. Anche perché le anomalie sulla storia di RaTG13 non finiscono qui.
Fino a maggio, infatti, eravamo tutti convinti che gli scienziati dell’Istituto di Virologia di Wuhan avessero sequenziato il genoma di RaTG13 soltanto dopo lo scoppio dell’epidemia. Dopo tutto, questo era quanto suggerito nell’articolo. Il 19 maggio, però, i ricercatori cinesi caricano sul portale NCBI alcune sequenze di RaTG13 e – colpo di scena – sono datate 2017/2018. Le sequenze, prodotte con sequenziamento Sanger, coprono gran parte del genoma, incluso il gene della proteina spike. E a confermare il sequenziamento del 2018 è la stessa Shi Zhengli, che rispondendo alle domande di Science ha dichiarato:
We detected the virus by pan-coronavirus RT-PCR in a bat fecal sample collected from Tongguan town, Mojiang county in Yunnan province in 2013, and obtained its partial RdRp sequence. Because the low similarity of this virus to SARS-CoV, we did not pay special attention to this sequence. In 2018, as the NGS sequencing technology and capability in our lab was improved, we did further sequencing of the virus using our remaining samples, and obtained the full-length genome sequence of RaTG13 except the 15 nucleotides at the 5’ end. As the sample was used many times for the purpose of viral nucleic acid extraction, there was no more sample after we finished genome sequencing, and we did not do virus isolation and other studies on it. Among all the bat samples we collected, the RaTG13 virus was detected in only one single sample. In 2020, we compared the sequence of SARS-CoV-2 and our unpublished bat coronavirus sequences and found it shared a 96.2% identity with RaTG13. RaTG13 has never been isolated or cultured.
Shi Zhengli, intervista a Science
Con queste parole, Shi Zhengli propone quindi una nuova versione dei fatti, rispetto a quella dichiarata nell’articolo pubblicato su Nature. RaTG13 non è stato sequenziato dopo aver confrontato il suo gene RdRp con quello di SARS-CoV-2, come ci era stato detto, bensì un paio di anni prima. Quello che hanno fatto quest’anno gli scienziati di Wuhan – dice la prof.ssa Shi – è stato confrontare l’intero genoma del nuovo coronavirus con i genomi dei coronavirus di pipistrello presenti nei loro archivi e non ancora pubblicati. Scopriamo inoltre che al genoma di RaTG13 mancano 15 nucleotidi all’inizio della sequenza, e che il campione originale raccolto nella miniera è stato ormai interamente consumato dai vari sequenziamenti. La misteriosa storia di RaTG13 dovrebbe dunque concludersi qui, non essendoci più materiale per fare altre analisi o esperimenti. Eppure, il 13 ottobre ecco un altro colpo di scena! Il genoma di RaTG13 caricato su NCBI viene improvvisamente aggiornato senza una spiegazione: appaiono finalmente i 15 nucleotidi mancanti, e 6 nucleotidi vengono cambiati. Il virus è stato sequenziato di nuovo? Hanno ritrovato vecchie sequenze che avevano inizialmente scartato per qualche motivo? Non lo sappiamo, perché Wuhan non ha mai pubblicato i dettagli delle analisi svolte su RaTG13, né tantomeno ha spiegato perché abbiano deciso di aggiornare il genoma.
Inutile dire che i complottisti di tutto il mondo vanno a nozze con questo continuo gioco di mezze verità, omissioni e sottintesi. Certo, pare piuttosto improbabile che SARS-CoV-2 sia il risultato di esperimenti fatti su RaTG13: dopotutto, tra i due genomi ci sono 1200 nucleotidi di differenza, una distanza difficilmente colmabile anche considerando l’evoluzione accelerata del laboratorio. Di fronte a tutte queste contraddizioni, però, non sembra poi così azzardato domandarsi, ad esempio, se in quella miniera non ci fossero magari altri coronavirus, più simili a SARS-CoV-2. Il dubbio è lecito, anche perché i ricercatori di Wuhan continuano a tenere nascosto un ricchissimo database specializzato in virus dei pipistrelli: il Wildlife-borne Viral Pathogen Database. Conteneva sequenze relative a 20mila campioni raccolti negli anni, alcune delle quali non ancora pubblicate. Oggi quel dataset non c’è più: sul web è rimasta solo la pagina che ne descriveva il contenuto, raggiungibile tramite la Wayback Machine di Internet Archive.
Aggiornamento 17/11/20 – C’è una grossa novità su RaTG13 e la storia della miniera. I ricercatori di Wuhan, che a febbraio avevano descritto il cugino di SARS-CoV-2 senza fornire dettagli sul suo ritrovamento, oggi hanno pubblicato un addendum all’articolo originale. In questa breve comunicazione, i ricercatori ammettono di essere stati coinvolti nelle indagini sulle misteriose polmoniti della miniera di Tongguan, nella contea di Mojiang. Avevano analizzato i campioni di siero di quattro pazienti, scrivono, ma non erano riusciti a identificare la causa della malattia. Ipotizzando che si trattasse di un nuovo virus, hanno visitato la miniera una o due volte all’anno dal 2012 al 2015, raccogliendo un totale di 1322 campioni. In questi campioni hanno identificato 293 coronavirus differenti, di cui 9 beta-coronavirus tutti di tipo SARS; uno di questi è RaTG13, il cui genoma è stato sequenziato per intero nel 2018, ad eccezione delle due estremità. In questo addendum viene quindi confermata la tempistica del sequenziamento fornita da Shi Zhengli alla rivista Science, in palese contraddizione con quanto scritto nello stesso articolo di Nature. Al fine di dimostrare che quelle persone non erano infette da SARS-CoV-2, i ricercatori hanno recentemente rianalizzato i campioni di siero alla ricerca di proteine del virus (curiosamente non hanno fatto PCR).
La comunità scientifica sarà felice di questa tardiva manifestazione di trasparenza, ma rimangono ancora molte domande. Ad esempio: come hanno fatto, poche settimane fa, ad aggiungere i 15 nucleotidi mancanti all’estremità del genoma di RaTG13, se il campione era esaurito? Perché nell’intervista a Scientific American di giugno, Shi Zhengli dice che la causa delle polmoniti era un fungo? I pazienti erano davvero negativi al test per gli anticorpi anti-SARS, come scritto nell’addendum, oppure erano positivi, come sostenuto nella tesi di laurea e anche in un’altra tesi di dottorato? Ma soprattutto: perché hanno atteso 9 mesi prima di rivelare al mondo che il ritrovamento di RaTG13 era legato alla storia di polmoniti misteriose (e letali) causate da un virus sconosciuto? Condivideranno finalmente con la comunità scientifica i campioni e le sequenze di questi 8 nuovi coronavirus di tipo SARS, oltre ai vecchi campioni di siero dei minatori malati?
Furina e pangolini
Se la pista dell’origine artificiale ci lascia con più domande che risposte, di certo quella dell’origine naturale non è prodiga di certezze. Sono usciti moltissimi articoli in questi mesi, volti principalmente a evidenziare che le caratteristiche uniche di questo coronavirus tanto uniche non sono. Nessuno di questi, però, è riuscito ad esempio a identificare l’animale ospite intermedio, anche se molti ci hanno provato. Tra marzo e aprile, sono stati pubblicati ben quattro articoli che sembravano suggerire un possibile ruolo dei pangolini nelle origini di SARS-CoV-2 (Lam et al., 2020; Xiao et al., 2020; Zhang et al., 2020; Liu et al., 2020). In quel periodo, eravamo letteralmente sommersi da articoli e notizie sui pangolini, tanto da far credere che avessimo finalmente trovato l’ospite intermedio. In realtà, però, pare che tutti e quattro questi studi abbiano analizzato i dati relativi agli stessi esemplari, un gruppo di pangolini malati sequestrati in un’operazione anti-bracconaggio avvenuta nel marzo 2019 nel Guangdong, e descritti per la prima volta a ottobre 2019 (Liu et al., 2019). D’altra parte, un’analisi fatta su oltre 300 pangolini sequestrati in Malesia dal 2009 al 2019 non ha trovato neppure un animale infetto da coronavirus, quindi è probabile che gli sfortunati esemplari del Guangdong fossero semplicemente un’eccezione alla regola. Inoltre, a quanto pare, gli articoli sui pangolini soffrirebbero di problemi legati a dati mancanti, campioni rianalizzati con nomi diversi, autori condivisi e il fatto bizzarro di essere stati tutti – eccetto uno – pubblicati in forma di preprint esattamente lo stesso giorno. La questione è piuttosto seria, tanto che Nature e PLoS Pathogens, due delle riviste che hanno pubblicato i lavori, hanno avviato delle indagini con gli autori per chiarire le obiezioni che sono state sollevate. Intanto, sulla pagina dell’articolo di Xiao et al. è già apparso un disclaimer piuttosto eloquente:
La storia dei pangolini è comunque interessante, perché il coronavirus di quegli animali salvati dai bracconieri condivide con il virus SARS-CoV-2 una parte piccola ma importantissima della proteina spike: il receptor binding domain (RBD), ossia la regione che fisicamente si lega al recettore umano. Il resto del genoma è troppo diverso, per questo quei pangolini non possono essere l’ospite intermedio che stiamo cercando. Tuttavia, quel coronavirus è l’unico, tra tutti quelli trovati nella fauna selvatica, ad avere una regione RBD quasi identica a quella di SARS-CoV-2: non ce l’ha neppure RaTG13, il virus cugino dei pipistrelli. Secondo alcuni, SARS-CoV-2 potrebbe essersi originato proprio dalla ricombinazione di un virus simile a RaTG13 con quello dei pangolini, in laboratorio (Segreto & Deigin, 2020) o in natura, in seguito alla coinfezione di uno stesso animale da parte dei due virus (Li et al., 2020). Altre analisi, invece, sembrano suggerire che questa ricombinazione non sia proprio avvenuta: non è stato SARS-CoV-2 ad acquisire dai pangolini la regione RBD che permette il legame con il recettore umano, ma al contrario è stato RaTG13 ad aver perso per strada, nel suo percorso evolutivo, la regione RBD originaria, che invece i suoi cugini hanno mantenuto (Boni et al., 2020). Tantissime ipotesi sul tavolo insomma, ma ancora poche certezze.
C’è un altro momento chiave per le ricerche sulle origini del virus: è quello in cui viene pubblicato lo studio che descrive il virus RmYN02, l’altro cugino di SARS-CoV-2, trovato anch’esso nello Yunnan (Zhou et al., 2020). La scoperta è importante perché si inserisce nel dibattito piuttosto acceso che si era scatenato attorno a una caratteristica molto particolare di SARS-CoV-2: la presenza sulla proteina spike di un sito di taglio per la furina. La furina è un’enzima umano che normalmente ha il compito di tagliare altre proteine umane, per attivarle e renderle funzionanti, ma alcuni virus hanno imparato a sfruttarla a proprio vantaggio: il taglio della furina, infatti, permette loro di entrare più facilmente in alcuni tipi di cellule. Non tutti però possiedono la sequenza segnale riconosciuta da questo enzima, e SARS-CoV-2 è l’unico coronavirus di tipo SARS ad averla (Coutard et al., 2020); nel caso specifico, sembra che aiuti il virus a infettare le cellule dei polmoni (Hoffman et al., 2020). Vista la rarità di questa sequenza di taglio, qualcuno ha ipotizzato che potesse essere stata inserita in laboratorio, così come d’altra parte era stato fatto con altri virus in passato (Follis et al., 2006; Yang et al., 2015). La scoperta di RmYN02, però, suggerisce che queste inserzioni possono avvenire anche in natura: il cugino di SARS-CoV-2 avrebbe infatti un’inserzione proprio nello stesso punto della proteina spike, anche se non tutti sono convinti (Segreto & Deigin, 2020). In ogni caso, questa inserzione non ha creato una sequenza di taglio per la furina, come è invece accaduto per il cugino più famoso.
Possibili scenari
Le contraddizioni degli scienziati di Wuhan suscitano inevitabilmente molti interrogativi. Ovviamente non esiste alcuna prova che i ricercatori cinesi abbiano qualcosa a che fare con le origini di SARS-CoV-2, ma le anomalie riscontrate in questi mesi certamente giustificano un’indagine seria e indipendente che ci aiuti finalmente a dare una risposta chiara alla domanda che tutti si fanno: da dove arriva il virus che ha stravolto le nostre vite? L’OMS sta cercando questa risposta insieme agli scienziati cinesi, incontrando qualche difficoltà, come scrive il New York Times. Il programma di lavoro concordato con la Cina è stato pubblicato il 5 novembre, e prevede, tra le altre cose: colloqui con i primi casi registrati a Wuhan; analisi degli animali e dei prodotti venduti al mercato di Huanan; consultazione delle vecchie cartelle cliniche; valutazione dei trend di polmoniti e sindromi influenzali nei mesi precedenti all’epidemia; analisi delle acque reflue e dei campioni di sangue raccolti prima di dicembre 2019. Il team dell’OMS svolgerà a distanza la maggior parte del lavoro, consultando risultati e documenti forniti dalla controparte cinese, ma è prevista anche una visita in Cina “al momento appropriato”. L’Istituto di Virologia di Wuhan, in ogni caso, non è neppure citato nel documento.
Come gran parte della comunità scientifica, anche l’OMS ritiene quindi che SARS-CoV-2 sia il risultato di una zoonosi, cioè del salto da un animale all’uomo. Gli scienziati convinti dell’origine naturale si basano sul rasoio di Occam: le precedenti epidemie ci hanno dimostrato che queste cose avvengono in natura, di conseguenza la spiegazione più semplice è che ciò sia avvenuto anche questa volta. A mio personale giudizio, però, si tratta di una presa di posizione discutibile, perché la fuga accidentale di un virus da un laboratorio è un evento raro, ma non impossibile. Inoltre, la presenza di un centro di ricerca specializzato in coronavirus, proprio nella città in cui è apparso SARS-CoV-2, è un fatto che non può essere ignorato, o liquidato come una curiosa coincidenza. Almeno, non prima di aver verificato su quali virus stavano lavorando i ricercatori di Wuhan. Scoprire le origini di SARS-CoV-2 è troppo importante per accontentarsi di speculazioni e probabilità “soggettive”: dobbiamo continuare a cercare. E forse, cercando, qualcosa troveremo.
Per esempio, potremmo trovare un virus molto simile a SARS-CoV-2 in qualche animale allevato nelle fattorie cinesi o commerciato dai bracconieri, come è accaduto con i pangolini. Qualche indizio per le ricerche potrebbe venire dagli animali che finora hanno dimostrato di essere suscettibili all’infezione: gatti, criceti, furetti, visoni. O forse scopriremo l’antenato di SARS-CoV-2 nei freezer di qualche ospedale cinese, a dimostrazione che il virus circolava da tempo senza che nessuno se ne fosse accorto. Oppure, per quanto questo scenario possa sembrare improbabile, troveremo le prove di esperimenti su SARS-CoV-2 nei laboratori dell’Istituto di Virologia di Wuhan, con conseguente crisi internazionale. Rimane purtroppo un’ultima possibilità, molto concreta: non scopriremo niente di tutto ciò, e la nostra domanda resterà per sempre senza una risposta.
Un ringraziamento speciale ad Alina Chan per la sua peer-review.