 Bisogna ammetterlo, gli organizzatori del Carnevale della Biodiversità conoscono il gusto della provocazione. Dopo “Le dimensioni contano”, “Nicchie estreme: ai confini della realtà” e “Alieni tra noi”, questa volta i partecipanti hanno affrontato un tema ancor più originale, che li ha costretti a spremere le meningi e a mettere sul campo tutta la loro creatività e fantasia. Il titolo di questa edizione del carnevale, tornato dopo una lunga pausa, ha infatti una connotazione fantascientifica piuttosto che scientifica: “Ho visto cose.. La biologia dei mondi fantastici”. Non appena l’ho letto, nella mia testa è scattato il collegamento con la biologia sintetica, un settore che ho iniziato a conoscere da vicino qualche mese fa, durante la mia visita all’ETH di Basilea. Che cosa c’è infatti di più fantastico di una biologia fabbricata dall’uomo, che in natura non esiste? Dopotutto, fare biologia sintetica significa realizzare molecole e organismi che non sono mai esistiti, e che tuttavia sarebbero potuti esistere se l’evoluzione avesse preso un altro corso. Fare biologia sintetica è un po’ come sbirciare in un mondo immaginario, un mondo che per la maggioranza di noi sarà popolato da draghi, fate e supereroi, ma che per un biologo molecolare potrebbe ospitare forme di vita ancora più bizzarre. Organismi diversi da quelli che conosciamo non solo per il loro aspetto o per il loro genoma, ma addirittura per il codice genetico in base al quale sono stati programmati.
Bisogna ammetterlo, gli organizzatori del Carnevale della Biodiversità conoscono il gusto della provocazione. Dopo “Le dimensioni contano”, “Nicchie estreme: ai confini della realtà” e “Alieni tra noi”, questa volta i partecipanti hanno affrontato un tema ancor più originale, che li ha costretti a spremere le meningi e a mettere sul campo tutta la loro creatività e fantasia. Il titolo di questa edizione del carnevale, tornato dopo una lunga pausa, ha infatti una connotazione fantascientifica piuttosto che scientifica: “Ho visto cose.. La biologia dei mondi fantastici”. Non appena l’ho letto, nella mia testa è scattato il collegamento con la biologia sintetica, un settore che ho iniziato a conoscere da vicino qualche mese fa, durante la mia visita all’ETH di Basilea. Che cosa c’è infatti di più fantastico di una biologia fabbricata dall’uomo, che in natura non esiste? Dopotutto, fare biologia sintetica significa realizzare molecole e organismi che non sono mai esistiti, e che tuttavia sarebbero potuti esistere se l’evoluzione avesse preso un altro corso. Fare biologia sintetica è un po’ come sbirciare in un mondo immaginario, un mondo che per la maggioranza di noi sarà popolato da draghi, fate e supereroi, ma che per un biologo molecolare potrebbe ospitare forme di vita ancora più bizzarre. Organismi diversi da quelli che conosciamo non solo per il loro aspetto o per il loro genoma, ma addirittura per il codice genetico in base al quale sono stati programmati.
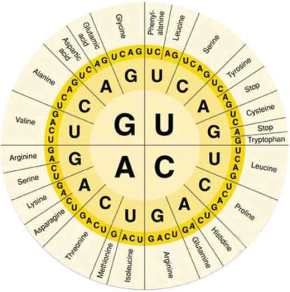 Ogni forma di vita di questo pianeta (a parte rare eccezioni) utilizza infatti lo stesso codice per trasformare in proteine funzionanti l’informazione genetica contenuta nel proprio DNA. E’ un codice a triplette, propriamente dette codoni, dove ogni tripletta di nucleotidi corrisponde a uno specifico aminoacido. Per esempio, se nel DNA c’è scritto AAA, la cellula inserirà nella catena della proteina nascente l’aminoacido lisina, e così via, tripletta dopo tripletta, finché la cellula non incappa in un codone di stop: lì il processo di traduzione termina e la nuova proteina può essere rilasciata. A dispetto dell’elevato numero di codoni possibili (64), gli aminoacidi comunemente a disposizione delle nostre cellule sono soltanto 20: significa che ogni aminoacido può essere codificato da più di un codone (tre codoni indicano un segnale di stop). Ma gli scienziati che si occupano di biologia sintetica non sono mai contenti di quello che l’evoluzione ha messo a punto nel corso di milioni di anni. In effetti, se solo potessimo convincere le cellule a utilizzare decine di altri aminoacidi diversi, potremmo ottenere proteine mai viste, anche con funzioni completamente nuove. Una prospettiva affascinante, non c’è dubbio, ma sorge un problema: se i 64 codoni sono già tutti prenotati, come si fa a cambiare il codice genetico senza che tutte le altre proteine, quelle naturali, vengano stravolte? Considerate che le cellule hanno le loro attività da portare avanti, e le proteine devono essere fatte bene, con tutti gli aminoacidi al posto giusto: se si prova ad alterare il significato di un solo codone, c’è il serio rischio che la cellula passi a miglior vita. Una soluzione al problema c’è, ma per capirla a fondo dovrete avere la pazienza di seguirmi mentre vi racconto più nel dettaglio il meccanismo della traduzione.
Ogni forma di vita di questo pianeta (a parte rare eccezioni) utilizza infatti lo stesso codice per trasformare in proteine funzionanti l’informazione genetica contenuta nel proprio DNA. E’ un codice a triplette, propriamente dette codoni, dove ogni tripletta di nucleotidi corrisponde a uno specifico aminoacido. Per esempio, se nel DNA c’è scritto AAA, la cellula inserirà nella catena della proteina nascente l’aminoacido lisina, e così via, tripletta dopo tripletta, finché la cellula non incappa in un codone di stop: lì il processo di traduzione termina e la nuova proteina può essere rilasciata. A dispetto dell’elevato numero di codoni possibili (64), gli aminoacidi comunemente a disposizione delle nostre cellule sono soltanto 20: significa che ogni aminoacido può essere codificato da più di un codone (tre codoni indicano un segnale di stop). Ma gli scienziati che si occupano di biologia sintetica non sono mai contenti di quello che l’evoluzione ha messo a punto nel corso di milioni di anni. In effetti, se solo potessimo convincere le cellule a utilizzare decine di altri aminoacidi diversi, potremmo ottenere proteine mai viste, anche con funzioni completamente nuove. Una prospettiva affascinante, non c’è dubbio, ma sorge un problema: se i 64 codoni sono già tutti prenotati, come si fa a cambiare il codice genetico senza che tutte le altre proteine, quelle naturali, vengano stravolte? Considerate che le cellule hanno le loro attività da portare avanti, e le proteine devono essere fatte bene, con tutti gli aminoacidi al posto giusto: se si prova ad alterare il significato di un solo codone, c’è il serio rischio che la cellula passi a miglior vita. Una soluzione al problema c’è, ma per capirla a fondo dovrete avere la pazienza di seguirmi mentre vi racconto più nel dettaglio il meccanismo della traduzione.
Fabbricare una proteina è un processo sofisticato nel quale intervengono diversi attori. Il ruolo del protagonista appartiene indubbiamente al ribosoma, un complesso macchinario composto da tre molecole di RNA e da più di 50 proteine. Spetta al ribosoma effettuare la sintesi vera e propria, andando a decodificare la sequenza nucleotidica dell’RNA messaggero in arrivo dal nucleo. I codoni vengono fatti scorrere, l’uno dopo l’altro, pronti per essere letti. Ed è qui che interviene l’altra molecola decisiva in questo processo, la chiave di lettura che consente al ribosoma di interpretare l’informazione portata dal messaggero e trasformarla in una catena di aminoacidi: sono i tRNA (o RNA transfer). Questi RNA di trasporto raggiungono il ribosoma e vanno ad appaiarsi con il codone su cui questo sta transitando. Se il codone si lega di buon grado all’anticodone portato dal tRNA, ecco che avviene la magia e il tRNA cede il suo prezioso carico: un aminoacido, che viene immediatamente trasferito alla catena proteica nascente. Il processo va avanti finché il ribosoma non incontra uno dei tre codoni di stop (UAA, UAG, UGA), che non riescono ad appaiarsi ad alcun tRNA. La traduzione funziona perché ogni codone può appaiarsi a un solo tRNA, e quel tRNA porterà sempre con sé lo stesso identico aminoacido. Già, ma una volta compiuto il loro dovere, che fanno i tRNA? Bussano alla porta degli enzimi amminoacil-tRNA sintetasi, che li ricaricano consegnando loro un nuovo aminoacido. Esistono 20 tipi diversi di questo enzima, ognuno competente per uno specifico aminoacido. Per cambiare il codice genetico e scriverne uno nuovo occorre dunque intervenire su tutti e tre questi componenti: il ribosoma, i tRNA e l’aminoacil-tRNA sintetasi. E’ quello che stanno cercando di fare all’Università di Cambridge, nel gruppo di ricerca guidato da Jason W. Chin. Il primo passo è stato creare un ribosoma alternativo a quello naturale, vediamo come hanno fatto.

![]() Chin e colleghi hanno fatto le cose in grande, e hanno deciso che il codice a triplette non bastava. Se proprio vogliamo scrivere un codice nuovo di zecca – hanno pensato – perché non farne uno con dei codoni di quattro lettere? Il vantaggio è evidente: si avrebbero a disposizione ben 256 codoni da riempire con gli aminoacidi più bizzarri. La prima cosa da fare per costruire un ribosoma del genere è impedire che il nostro esperimento molecolare interferisca con il ribosoma naturale, che deve continuare a svolgere correttamente il proprio lavoro. Sembrerà un accorgimento banale, ma non lo è affatto: per riuscire in questo compito, i ricercatori inglesi hanno dovuto mettere a punto un disegno sperimentale molto astuto, sfruttando la cosiddetta “sequenza di Shine-Dalgarno”. Questa sequenza si trova all’inizio degli RNA messaggeri batterici, e funziona come segnale di riconoscimento per i ribosomi. L’idea di Chin e colleghi era di cambiare in qualche modo questa sequenza, in modo da realizzare dei messaggeri artificiali che fossero invisibili ai ribosomi. Hanno quindi prodotto una serie di possibili varianti di questa sequenza e l’hanno inserita in un gene ad hoc, che codificava per due proteine fuse insieme (capirete presto perché): la prima proteina neutralizzava l’effetto di un antibiotico (cloramfenicolo), la seconda era tossica in presenza di 5-fluorouracile (5-FU). Somministrando quest’ultima molecola, il team di Chin ha dato inizio alla strage: tutte le cellule che nel messaggero artificiale avevano una sequenza di Shine-Dalgarno riconosciuta dal ribosoma producevano la proteina tossica e morivano. I ricercatori erano così riusciti a scoprire quali sequenze di Shine-Dalgarno non piacevano ai ribosomi naturali. Sono state queste sequenze il primo importante passoverso la creazione di un sistema di traduzione alternativo, che non interferisse con quello originale. Ma ora bisognava realizzare un ribosoma nuovo che quelle sequenze, invece, fosse in grado di riconoscerle. I ricercatori hanno fatto anche questo. Hanno fatto sintetizzare alle cellule dei ribosomi leggermente differenti rispetto a quello di partenza, e hanno poi selezionato quelli che casualmente riuscivano a riconoscere una delle sequenze di Shine-Dalgarno alternative (per fare lo screening questa volta hanno usato l’antibiotico cloramfenicolo). Per sopravvivere all’antibiotico le cellule dovevano avere un gran colpo di fortuna: l’unica salvezza per loro era possedere la coppia messaggero/ribosoma che consentiva la produzione della proteina salvavita. Beh, qualcuna ce l’ha fatta: su un miliardo di combinazioni testate, tre hanno avuto successo. E con questo risultato, i ricercatori hanno potuto sperimentare liberamente sul loro ribosoma alternativo, senza intaccare il normale processo della traduzione. Ora potevano iniziare a fare esperimenti in tranquillità, nel tentativo di creare un nuovo codice della vita basato su codoni di quattro lettere.
Chin e colleghi hanno fatto le cose in grande, e hanno deciso che il codice a triplette non bastava. Se proprio vogliamo scrivere un codice nuovo di zecca – hanno pensato – perché non farne uno con dei codoni di quattro lettere? Il vantaggio è evidente: si avrebbero a disposizione ben 256 codoni da riempire con gli aminoacidi più bizzarri. La prima cosa da fare per costruire un ribosoma del genere è impedire che il nostro esperimento molecolare interferisca con il ribosoma naturale, che deve continuare a svolgere correttamente il proprio lavoro. Sembrerà un accorgimento banale, ma non lo è affatto: per riuscire in questo compito, i ricercatori inglesi hanno dovuto mettere a punto un disegno sperimentale molto astuto, sfruttando la cosiddetta “sequenza di Shine-Dalgarno”. Questa sequenza si trova all’inizio degli RNA messaggeri batterici, e funziona come segnale di riconoscimento per i ribosomi. L’idea di Chin e colleghi era di cambiare in qualche modo questa sequenza, in modo da realizzare dei messaggeri artificiali che fossero invisibili ai ribosomi. Hanno quindi prodotto una serie di possibili varianti di questa sequenza e l’hanno inserita in un gene ad hoc, che codificava per due proteine fuse insieme (capirete presto perché): la prima proteina neutralizzava l’effetto di un antibiotico (cloramfenicolo), la seconda era tossica in presenza di 5-fluorouracile (5-FU). Somministrando quest’ultima molecola, il team di Chin ha dato inizio alla strage: tutte le cellule che nel messaggero artificiale avevano una sequenza di Shine-Dalgarno riconosciuta dal ribosoma producevano la proteina tossica e morivano. I ricercatori erano così riusciti a scoprire quali sequenze di Shine-Dalgarno non piacevano ai ribosomi naturali. Sono state queste sequenze il primo importante passoverso la creazione di un sistema di traduzione alternativo, che non interferisse con quello originale. Ma ora bisognava realizzare un ribosoma nuovo che quelle sequenze, invece, fosse in grado di riconoscerle. I ricercatori hanno fatto anche questo. Hanno fatto sintetizzare alle cellule dei ribosomi leggermente differenti rispetto a quello di partenza, e hanno poi selezionato quelli che casualmente riuscivano a riconoscere una delle sequenze di Shine-Dalgarno alternative (per fare lo screening questa volta hanno usato l’antibiotico cloramfenicolo). Per sopravvivere all’antibiotico le cellule dovevano avere un gran colpo di fortuna: l’unica salvezza per loro era possedere la coppia messaggero/ribosoma che consentiva la produzione della proteina salvavita. Beh, qualcuna ce l’ha fatta: su un miliardo di combinazioni testate, tre hanno avuto successo. E con questo risultato, i ricercatori hanno potuto sperimentare liberamente sul loro ribosoma alternativo, senza intaccare il normale processo della traduzione. Ora potevano iniziare a fare esperimenti in tranquillità, nel tentativo di creare un nuovo codice della vita basato su codoni di quattro lettere.
A onor del vero, in passato erano già stati condotti esperimenti in cui si cercava di introdurre aminoacidi non naturali sfruttando le quadriplette al posto delle triplette, ma con i ribosomi naturali questa operazione risultava complicata: i tRNA speciali in grado di appaiarsi a codoni di quattro lettere fanno fatica a entrare nel ribosoma, e d’altra parte ogni tentativo di aumentare l’efficienza di traduzione rischiava di danneggiare in modo letale tutte le altre proteine. Con il ribosoma alternativo messo a punto dal gruppo di Jason W. Chin non c’era più questo pericolo. I ricercatori hanno quindi testato oltre un miliardo di ribosomi leggermente diversi tra loro, con l’obiettivo di trovarne uno che fornisse un alloggio più comodo per i tRNA speciali. Ancora una volta hanno costretto le cellule a fabbricare la proteina che dava resistenza al cloramfenicolo, ma in questo caso il gene che la codificava aveva in un certo punto un codone di quattro lettere, che poteva essere letto solo se il ribosoma riusciva ad ospitare un apposito tRNA speciale introdotto dai ricercatori. E’ bastato aggiungere un po’ di antibiotico per fare fuori tutte le cellule prive di questo superpotere e scovare il ribosoma in grado di leggere i codoni da quattro lettere tanto quanto quelli da 3: lo hanno chiamato Ribo-Q1.
 Finalmente abbiamo dunque il ribosoma che fa per noi. Il problema che hanno dovuto affrontare i ricercatori di Cambridge riguardava a questo punto i tRNA. Posso anche creare dei tRNA speciali che si appaino alle quadriplette portando con sé degli aminoacidi non naturali, ma quegli aminoacidi qualcuno deve fornirli. Come ho scritto all’inizio, questo compito normalmente è svolto dagli enzimi amminoacil-tRNA sintetasi, ma se voglio usarli per ricaricare anche i miei tRNA speciali devo stare bene attento che non vadano a interferire con i tRNA normali. Per questi enzimi vale un po’ lo stesso discorso fatto per i ribosomi: il nuovo sistema di traduzione deve essere alternativo a quello naturale in tutto e per tutto, o in termini più tecnici deve essere ortogonale. Fortunatamente, la natura ci viene in soccorso, aiutandoci a risolvere almeno parzialmente il problema. Si dà il caso, infatti, che alcuni microrganismi produttori di metano abbiano sviluppato delle coppie sintetasi/tRNA che, inserite nel batterio Escherichia coli, non interferiscono con il suo processo di traduzione originario. Sfruttando questa proprietà, i ricercatori sono riusciti a far inserire al ribosoma Ribo-Q1 tutta una serie di aminoacidi non naturali.
Finalmente abbiamo dunque il ribosoma che fa per noi. Il problema che hanno dovuto affrontare i ricercatori di Cambridge riguardava a questo punto i tRNA. Posso anche creare dei tRNA speciali che si appaino alle quadriplette portando con sé degli aminoacidi non naturali, ma quegli aminoacidi qualcuno deve fornirli. Come ho scritto all’inizio, questo compito normalmente è svolto dagli enzimi amminoacil-tRNA sintetasi, ma se voglio usarli per ricaricare anche i miei tRNA speciali devo stare bene attento che non vadano a interferire con i tRNA normali. Per questi enzimi vale un po’ lo stesso discorso fatto per i ribosomi: il nuovo sistema di traduzione deve essere alternativo a quello naturale in tutto e per tutto, o in termini più tecnici deve essere ortogonale. Fortunatamente, la natura ci viene in soccorso, aiutandoci a risolvere almeno parzialmente il problema. Si dà il caso, infatti, che alcuni microrganismi produttori di metano abbiano sviluppato delle coppie sintetasi/tRNA che, inserite nel batterio Escherichia coli, non interferiscono con il suo processo di traduzione originario. Sfruttando questa proprietà, i ricercatori sono riusciti a far inserire al ribosoma Ribo-Q1 tutta una serie di aminoacidi non naturali.
Missione compiuta dunque? Non proprio. In natura esistono solo due coppie sintetasi/tRNA realmente ortogonali al sistema di traduzione di E. coli: sono le uniche che non disturbano la normale attività di traduzione, e la sola cosa che posso fare è cambiare i due aminoacidi associati a queste due coppie. Questo significa che è impossibile inserire in una proteina più di due aminoacidi non naturali! Ecco dunque la vera sfida per le prossime ricerche: se vogliamo produrre polimeri completamente non naturali, bisogna escogitare strategie per sviluppare nuove coppie sintetasi/tRNA disponibili ad accettare altri aminoacidi. Se i biologi sintetici riusciranno nell’impresa, in futuro potremmo fare esperimenti molto interessanti e scoprire che, con qualche aminoacido in più a disposizione, la vita potrebbe evolvere funzioni nuove e imprevedibili, funzioni che per la biologia naturale sono fuori portata. Ma qui si entra nel regno della fantascienza, anzi, si entra nella biologia dei mondi fantastici. Trovate gli altri contributi al Carnevale della Biodiversità sul blog Mahengechromis. Buona lettura!
Wang, K., Schmied, W., & Chin, J. (2012). Reprogramming the Genetic Code: From Triplet to Quadruplet Codes Angewandte Chemie International Edition, 51 (10), 2288-2297 DOI: 10.1002/anie.201105016
tRNA-sintetasi, quadriplette e ribosomi ortogonali: nel mondo di Chin o della serie “viaggio al centro della sintesi sintetica” – ma neanche Kirk e l’Enterprise!
Una bella “bio-officina” creata con la “selezione innaturale”, ma adesso gli obiettivi?
La selezione casuale per vedere cosa capita di interessante potrebbe non essere una strada percorribile in tempi umani: la selezione di queste funzioni di sintesi è stata fatta mirando un obiettivo preciso: la macchina di codifica per codoni a 4 basi, ma come si fa ad avere obiettivi proteici le cui funzioni sono sconosciute? (fare catene di mRNA a caso e stare a vedere?)
Il tuo dubbio è legittimo! Però la biologia sintetica funziona ancora un po’ così, a volte addirittura è solo un esercizio tecnico per affinare delle tecniche e dire “ok, questo possiamo farlo”. Lo scopo di questo lavoro è di produrre strumenti utili che poi verranno messi a disposizione di chi vorrà inventarsi qualche applicazione. Ci sono gruppi che studiano come si relaziona la struttura di una proteina alla sua funzione, in ambito medico o industriale.. Magari un giorno faranno qualche scoperta che per tradursi in applicazione avrà proprio bisogno di questa “bio-officina”.
Che tu sappia la strada della nanotecnologia a scopo nanostrutture (e in futuro nanorobotica) prende in considerazione la produzione a mezzo ribosomi o gli studi sono a esclusivo beneficio si sintesi proteica a uso biologico?
PS i gruppi di studio che citi sono quelli realitivi alla proteomica?
Sì, proteomica. Quanto alle nanotecnologie non saprei, forse c’è qualcosa ma di sicuro non è una applicazione di routine. Devo ammettere però che le nanotecnologie non sono il mio campo!
Da quello che dici qui penso che presto avremo convergenza nanotecnologia-genetica: la nanotecnologia ha bisogno di autoreplicazione e la vita già lo fa, non vedo alternative logiche se non cominciare da lì (e Venter mi pare confermi l’idea), dopotutto la scienza dell’artificiale ha sempre cominciato come imitazione della natura, no?
Comunque forse hai ragione: è troppo presto per vedere già delle sinergie interdisciplinari … o forse no visto che hanno già costruito un motore molecolare: perché non farne costruire miliardi da batteri?
Sì sicuramente in futuro sarà così! Può anche darsi che qualcuno ci stia già lavorando adesso, sicuramente le cellule sono molto efficienti nel produrre proteine, poi la fattibilità dipende molto dalla specifica applicazione, dalla complessità della cosa che vuoi far fare ai batteri eccetera.. Ma le cose si stanno muovendo sicuramente nella direzione di una convergenza.
Da laureata in biotecnologie e studentessa magistrale di bionanomateriali, posso affermare che la ricerca di questi ultimi dieci, forse quindici anni, si è orientata sull’unione delle nanotecnologie con la biologia. In primo luogo, le nanotecnologie cercano di imitare ciò che da sempre la natura fa magistralmente. Un esempio tra tutti è l’autoassemblaggio della doppia elica del DNA. Sappiamo che esso si basa sulla formazione spontanea e reversibile di legami a idrogeno tra le basi di due filamenti complementari. Si è cercato quindi, prendendo spunto da questo processo naturale, di creare artificialmente delle strutture, che in risposta a stimoli di natura chimica o fisica, e quindi in modo del tutto controllabile dall’operatore, dessero luogo a legami tra componenti chimicamente complementari, con conseguente formazione di strutture supramolecolari. Tali strutture possono avere poi svariate applicazioni e funzioni in tutti i campi. Altro motivo di sposalizio tra le due scienze, è il fatto che la nanotecnologia può avere applicazioni biologiche, ad esempio la “drug delivery”, cioè la somministrazione mirata di farmaci a cellule tumorali, mediante l’iniezione per via parentale di nanoparticelle coniugate ad un farmaco e ad un fattore di crescita (Epidermal Growth Factor), che si lega in modo specifico alle cellule tumorali che esprimono sulla loro superficie il corrispondente recettore (Epidermical Growth Factor Receptor). Il rilascio del farmaco anche in questo caso avviene grazie a uno stimolo (es. pH acido proprio delle cellule tumorali). Ultimo aspetto interessante è che le nanotecnologie fanno uso di strutture biologiche. Ad esempio, avviene la biomineralizzazione di capsidi virali con ossidi di metalli, per creare degli efficienti nanocontainers e nanoreattori. Negli anni più recenti, è stata investigata anche la possibilità di utilizzare microorganismi per la produzione di nanostrutture, come nanoparticelle, grazie alla proprietà di molti batteri e funghi di ridurre chimicamente i metalli.
Detto questo, mi scuso per essere stata così prolissa e aver sfruttato forse inappropriatamente lo spazio a disposizione, però visto che si parlava di nanotecnologie e biologia, mi sono sentita di riportare qualche conoscenza, recentemente acquisita, su argomenti che mi piacciono molto. Mi complimento poi per il blog, in cui oggi, per la prima volta casualmente mi sono imbattuta, ma che mi sono promessa comincerò a seguire. Buon lavoro e buon proseguimento!