Genome Biology quest’oggi ha pubblicato una review molto interessante con la firma di Lincoln Stein, dell’Ontario Institute for Cancer Research di Toronto. L’articolo è un’analisi molto precisa e puntuale su quanto sta avvenendo nel mondo della biologia dopo l’avvento nel 2005 delle tecnologie di sequenziamento next-generation, e si sofferma in particolare su un aspetto che viene a mio avviso troppo spesso trascurato pur rivestendo invece un’importanza fondamentale: le risorse computazionali necessarie per far fronte a moli di dati sempre crescenti.
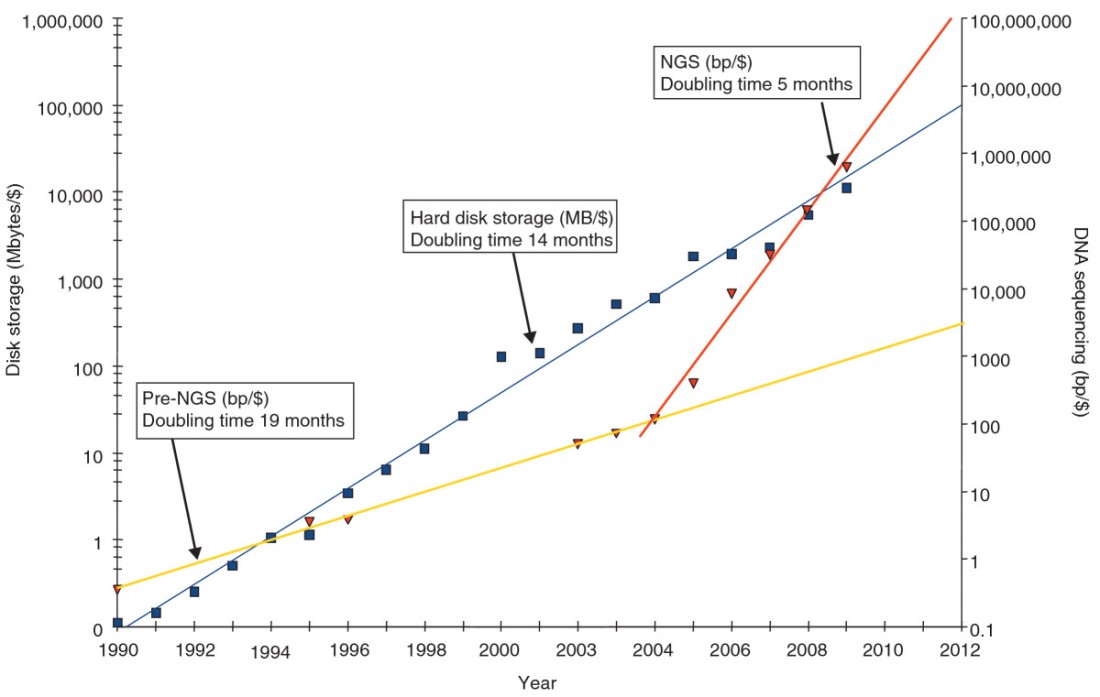
Stein ha registrato l’andamento negli ultimi 20 anni dei costi relativi allo stoccaggio di dati su Hard Disk e lo ha messo a confronto con i costi di produzione di sequenze genomiche. Il risultato, notevole e al tempo stesso preoccupante, è riportato nel grafico logaritmico sottostante. Dal 1990 a oggi, il costo di stoccaggio di un byte di dati si è dimezzato ogni 14 mesi, in buon accordo con la legge di Kryder, la quale sostiene che la capacità di un disco rigido a parità di prezzo raddoppia approssimativamente ogni anno. Tutto sembrerebbe andare liscio anche guardando la linea relativa ai costi del sequenziamento Sanger vecchia maniera: se non fossero state inventate le tecnologie di nuova generazione le due rette non si sarebbero mai incontrate. Ma osserviamo cosa accade nel 2005: con l’apparizione sul mercato delle macchine high-throughput (454, Solexa/Illumina, ABI SOLiD), i costi di un progetto di sequenziamento iniziano a precipitare molto più rapidamente. Ogni 5 mesi con la stessa cifra è possibile produrre il doppio delle sequenze! Non c’è garanzia che il trend continui in modo indefinito, ma tutto lascia pensare che le aziende produttrici (Illumina, Pacific Biosystems, Helicos, Ion Torrent) continueranno in questa direzione, rifinendo e migliorando delle tecnologie già anni luce lontane dalle capacità di un sequenziatore Sanger.
Fino a qualche anno fa si poteva benissimo mantenere i medesimi dati non solo in un archivio centrale (GenBank e affini), ma anche in database secondari e addirittura scaricarli per intero sui cluster di tutti i gruppi di bioinformatica sparsi nel mondo che volevano analizzarli. Dopotutto, la maggior parte dei dataset genomici potevano stare su un DVD (il genoma umano occupa 3 Gb). Visti i dati di Stein, però, sembra sarà presto necessario abbandonare questo modello di gestione dei dati per passare a uno più moderno, quello che va sotto il nome di cloud computing. Nell’idea dell’autore, per altro ragionevolmente condivisibile, non ci saranno alternative se non quella di far gestire a un grande service provider tutte le infrastrutture informatiche necessarie per la memorizzazione dei dati, oltre alla capacità computazionale richiesta per il loro utilizzo. Le risorse saranno così condivise tra tutti i database principali in un’unica grande nuvola, alla quale si potrà accedere mediante virtual machines.
L’ultima questione sollevata da Stein riguarda la banda di rete per la trasmissione dei dati: con le velocità attualmente a disposizione delle università e dei centri di ricerca, trasferire 100 gigabytes di sequenze richiederebbe alcuni giorni. In questo caso, il problema potrebbe essere risolto tornando a una vecchia prassi utilizzata anni fa dalla Protein Data Bank, la quale consentiva di depositare strutture proteiche spedendo i dati via posta tradizionale.
Stein LD: The case for cloud computing in genome informatics, Genome Biology 2010, 11:207
Un pensiero riguardo “Sequenze tra le nuvole”